直觉上,基因敲除(knockout, KO)仿佛是比基因抑制(knockdown, KD)更“彻底”的一种破坏基因表达的方法。那是否意味着,KO一个基因所得到的的表型一定比KD更加明显呢?答案是不一定。
生物学家对基因功能的研究,往往需要将“该基因正常发挥作用”的个体与“该基因表达异常”的个体进行对比,从异常的表型(phenotype)中推断这个基因的功能。早期,人们只能从自然界随机出现、或人为大规模诱导突变得到表型异常的个体中选择研究对象,鉴定到底是哪个或哪些基因异常(genotype)引起这个表型。突变常常是随机的,即便可以通过一些“筛选”富集到与所研究的功能相关的突变,但这一过程仍然很大程度上是盲人摸象。这种先看到表型,再去研究和鉴定背后主导基因的研究方法被称为正向遗传学(Forward genetics)。
后来,拥有了越来越多可以直接对基因进行干扰或破坏,继而观察这种干扰或破坏引起了哪些异常表型的工具,这样的研究被称为反向遗传学(Reverse genetics)[1]。在发育生物学中,人们常使用一种叫Morpholino(MO)的工具,这是一种核酸类似物,“其基本原理是把核苷酸上面的五碳糖环用一个吗啉环(morpholino)取代,同时对原有的磷酸基团也做了改变。使得这样一个DNA分子类似物可以以碱基互补配对的方式同RNA和DNA单链结合,但是由于结构的改变,使得整个分子不带有任何电荷,因此无法被任何酶所识别,包括DNase和RNase. 使得这类物质在细胞内有着极强的稳定性。”
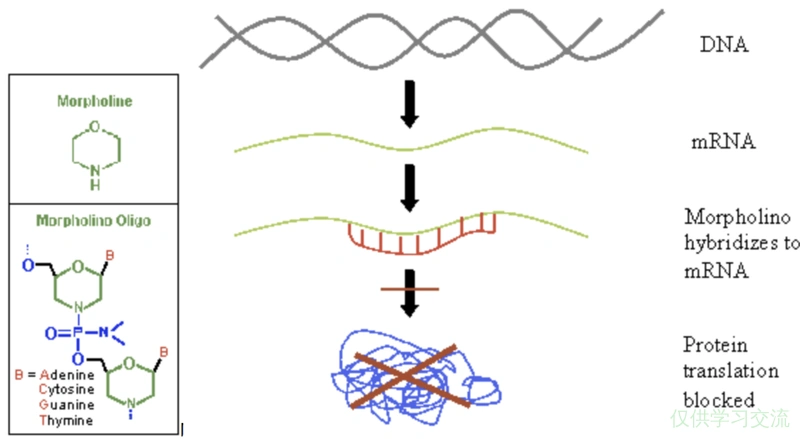
Morpholino结合目标mRNA后,阻碍一切酶对mRNA的结合,达到抑制表达效果,如结合在mRNA的上游会阻碍核糖体40S亚基向mRNA的结合,结合在splice sites会阻碍剪接,结合在miRNA靶点抑制miRNA的功能等[3]。Morpholino在发育生物学中被广泛应用,最常见的用法是直接向胚胎中注射MOs来KD某个基因。斑马鱼在桑椹胚时期的各个细胞之间形成extensive cytoplasmic bridges,所以可以通过注射Morpholino对全胚胎进行操作。在同阶段的爪蟾胚胎中,细胞质分裂是完全的,带来的一个优势是,可以在二细胞期对一个细胞注射Morpholino,这样就得到一个细胞作为实验组,另一个作为对照组[4]。
如此一来,发育生物学家获得了越来越多重要基因KD后的表型,也借此阐明了许多发育过程背后的分子基础。然而,MOs并不是完美的,其off-target效应一直是一个很大的问题,即我们看到的表型可能并不是因为目的基因被敲降导致的,而是因为一个与课题完全不相干的基因被意外地敲降而造成。一个经验是“25-mer的MO中有4个错配依然可以在高浓度下达到相同的效果”[5]。
再后来,更牛逼的工具来了,TALEN和CRISPR/Cas9等真正实现了彻底从基因组上“敲除”一个基因。新工具如此简便好用,而MOs的off-target风险一直不可忽视,业内人士开始议论,是否要放弃MOs的主流地位,甚至在期刊审核时能用CRISPR/Cas9的地方必须要求使用CRIPSR/Cas9,而不能只提供MOs的数据[6]。
暂且不论圈子内是否就Morpholino的去留达成共识,使用TALEN和CRISPR/Cas9敲除基因的数据不可抵挡地在积累。于是,对于同一个基因,既有通过传统MOs(或Anti-sense Oligonucleotides[3]等)工具KD的数据,也有使用CRISPR/Cas9等KO的数据,这两套结果自然会被放在一起对比。理想情况下,彻底沉默掉一个基因,和敲除一个基因,结果都是造成该基因表达的蛋白质不能正常产生并发挥作用,那么引起的宏观个体水平上的表型就应该是一样的。哪怕不一样,KO从源头上改变DNA序列,也应该更“彻底”,产生更严重的phenotype。
令人咋舌的是,事实跟我们预计的恰好相反,KO得到的表型往往比KD得到的表型更“轻微”,更有甚者几乎看不到与野生型的差别。这样的例子如此之多,从爪蟾、斑马鱼、小鼠到人中都有这样的事情被文献报道出来。
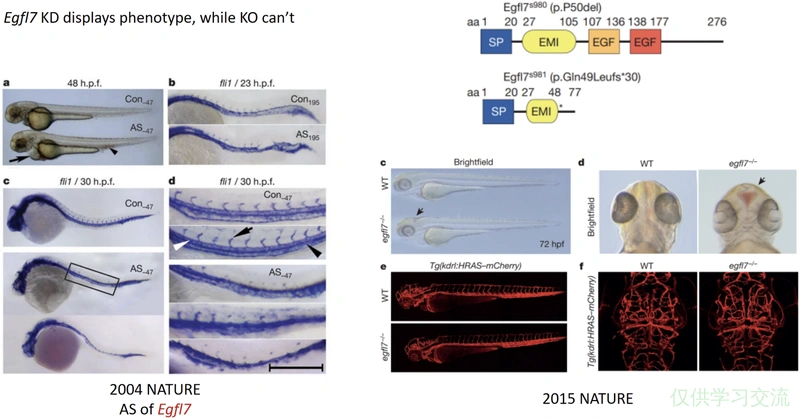
举个例子,上图中Egfl7是个在斑马鱼心血管发育中起作用的基因,左边是用Anti-sense Oligonulceotides (ASO)对这个基因进行KD,可以看到AS组对比Con组(control)发生明显的血管发育异常(说人话就是你看AS里那个染成蓝色的血管比Con细了,而且侧枝都消失了,咱不懂,但也能看出来是个严重的异常表型),右图是用CRIPSR/Cas9对Egfl7进行KO,可以看到mCherry标记的区域跟WT根本看不出来差异,唯一的一点点区别是右上角egfl7-/-的脑门那里有一点红,但对比左图的表型就轻微得多了[7]。
究竟是什么原因,导致事实跟想象不一样呢?大致猜想几种可能[8]:
- KD的靶标是mRNA,而KO的靶标是基因组。而对于胚胎而言,直到MBT(Mid-Blastula Transition,中囊胚期转换)之后,合子基因才开始表达,之前的母源mRNA的表达并不会受到KO的影响,因此,如果胚胎内拥有一个基因的母源mRNA,KO的表型就会更弱。
- 如前所述,MOs的脱靶效应。
- KO时造成的突变并没有完全使它失活,而是保留一部分活性,成为hypomorphic allele [9].
- 因为某些不可知的原因,mutant中发生了一些遗传补偿(genetic compensation),而morphant[10].中没有发生。
Didier Stainier是马普所的一名(大佬)研究员,是国际一个研究zebrafish的什么什么协会的主席,作为leader,有责任回答一下年会上会员们的争议问题[11],于是找个几个研究牲对这个问题开展了研究[12]。
简而言之,他发现他找的那几个基因(表现出KD表型比KO更明显的)——有一个共同的特点,在它们被CRISPR/Cas9 KO后,基因组中与它们序列相似的基因(最相似的那部分基因就是它的paralogs—旁系同源基因)的表达量会上升,因为序列相似在概率上功能也相似,所以就“回补”了一些该基因KO造成的表型异常,如下图的Egfl7和emilin家族。

他还发现,同一个基因用CRISPR/Cas9 KO,有的mutant的paralogs表达上调,有的则一点都不上调。区别在于,前者被KO的基因的mRNA被大量decay,后者没有。
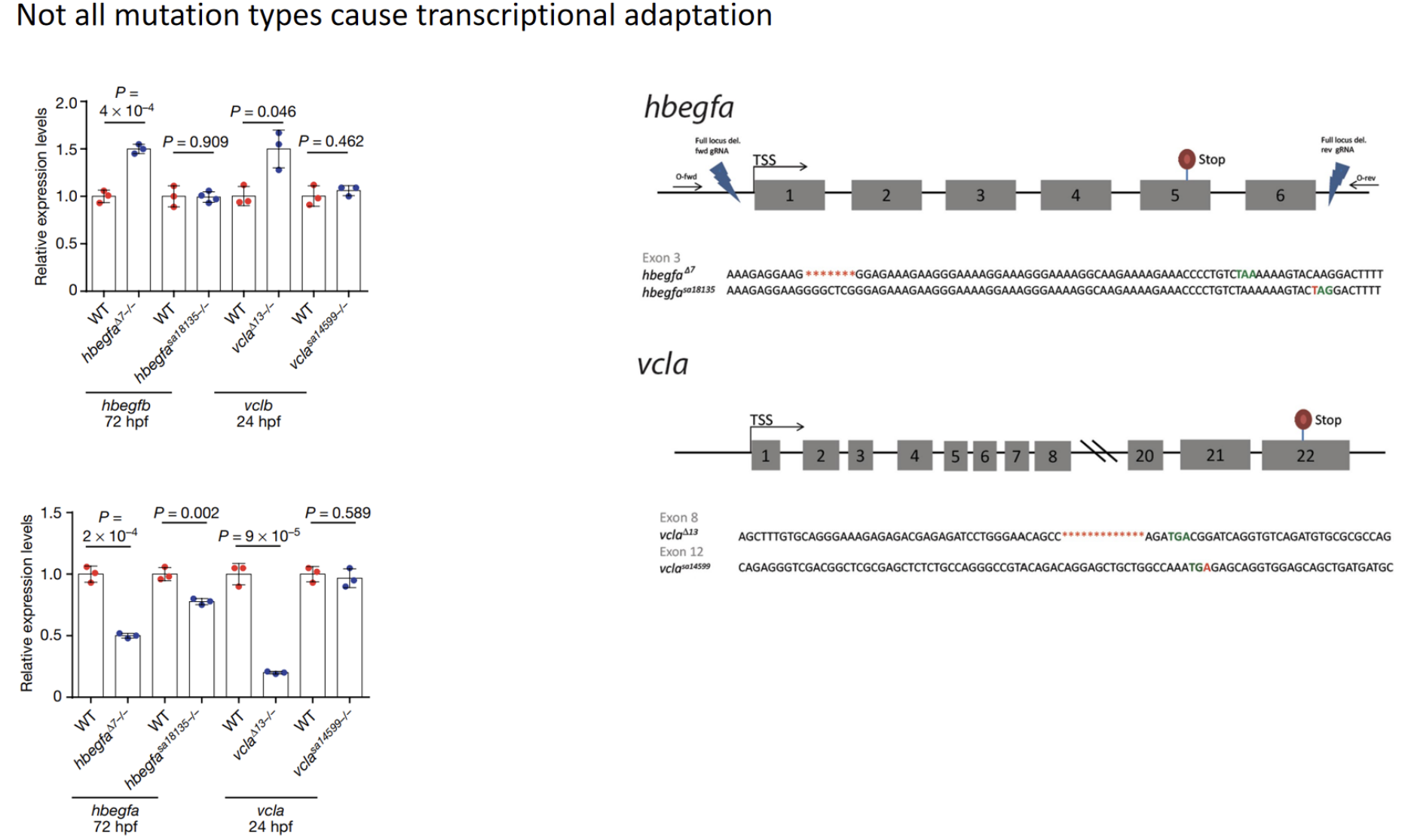
所以他的结论是——KO在mutant中产生一条无功能的mRNA,被RNA surveillance系统监测到以后被细胞主动decay掉,这个decay过程诱发与它序列相似的基因表达量上调,这些基因中如果包括一些功能也类似的基因,就补偿了KO造成的表型异常。他还发明了一个词来描述他发现的这个过程——转录适应(Transcriptional adaptation)[14]。

这个研究至少是部分解释了KO表型轻于KD的现象,但显然是不完全的。Transcriptional adaptation只是Compensation的一种形式,生物还有更多的compensation的机制交织在一起。
早在这篇文章发表以前,人们早就怀疑可能是paralogs上调对mutant的表型进行了compensation. 2017年有篇文章题目就叫“Comparison of Zebrafish tmem88a mutant and morpholino knockdown phenotypes”[15],比较斑马鱼tmem88a的mutant和morpholino KD的表型。作者也是发现mutant的phenotype显著弱于morphant,他们也怀疑是不是有paralogs上调了,但做了实验发现并没有。在conclusion中,他们把这差异归结到MOs的脱靶效应上。但显然这仍然是猜测,compensation远比我们想象的要复杂。

总之,技术都有优缺点,MOs至今仍然地位不可撼动,CRISPR/Cas9如日中天的同时,诸如compensation之类的缺点也暴露出来。当然就算不谈这些,knockdown也在很多场景下是不可代替的。咱们好好学深学透、活学活用才是真。
[1] Didier Stainier在多篇文章中认为KD算不上反向遗传学,只有KO才算,不过我没看到他解释为什么,也许是学界公认。
[2] 什么是morpholino?https://www.ncbi.nlm.nih.gov/probe/docs/techmorpholino/
[3] 被Morpholino结合的mRNA不会被降解,所以当转录出来的mRNA数量越来越多时,是有可能结合全部Morpholino的,不过,绝大多数情况下,都会注射过量的Morpholino. 当然,根据实验所需,也可以控制MOs实现一定“程度”的KD。Anti-Sense Oligo(ASO)也是研究胚胎发育中常用的工具。ASO(anti-sense oligonucleotides)是一段与mRNA互补的DNA序列,结合mRNA后诱导其被RNaseH降解。ASO不如Morpholino那么稳定,一般会加点修饰防止过快降解。在研究发育时,可以通过控制ASO的量,实现只对MBT之前母源mRNA进行降解,而在MBT后,ASO降解,合子基因组产生的mRNA不受抑制。
[4] Bestman, J.E., Cline, H.T. (2020). Morpholino Studies in Xenopus Brain Development. In: Sprecher, S. (eds) Brain Development. Methods in Molecular Biology, vol 2047. Humana, New York, NY. https://doi.org/10.1007/978-1-4939-9732-9_21
[5] Eisen JS, Smith JC. Controlling morpholino experiments: don’t stop making antisense. Development. 2008 May;135(10):1735-43. doi: 10.1242/dev.001115. Epub 2008 Apr 9. PMID: 18403413.
设置充分的对照,以最大程度排除off-target对实验造成的干扰:
- 对照组设计
- 本物种没有的“外源”基因的MOs(尽量选择和靶基因更像的)作为阴性对照
- 设计MO前使用Blast,尽量不选择跟其他基因有相似序列的区域
- 使用不完美配对的MOs
- 使用5个核苷酸错配的MO作为对照
- 使用不同MOs靶向同一个基因
- Rescue
- 这是最可靠的control
- 以一种对MO免疫的形式回补这个被MO敲降的基因
- 对于普遍高表达的基因来说,rescue相对容易;但如果该基因原本的表达量是精确控制的,就很难人为rescue.
[6] 这段是我轮转老板(作为业内老人)口述的发生过的争议。
[7] Rossi A, Kontarakis Z, Gerri C, Nolte H, Hölper S, Krüger M, Stainier DY. Genetic compensation induced by deleterious mutations but not gene knockdowns. Nature. 2015 Aug 13;524(7564):230-3. doi: 10.1038/nature14580. Epub 2015 Jul 13. PMID: 26168398.
[8] Stainier DYR, Raz E, Lawson ND, Ekker SC, Burdine RD, Eisen JS, Ingham PW, Schulte-Merker S, Yelon D, Weinstein BM, Mullins MC, Wilson SW, Ramakrishnan L, Amacher SL, Neuhauss SCF, Meng A, Mochizuki N, Panula P, Moens CB. Guidelines for morpholino use in zebrafish. PLoS Genet. 2017 Oct 19;13(10):e1007000. doi: 10.1371/journal.pgen.1007000. PMID: 29049395; PMCID: PMC5648102.
[9] What is “Hypomorphic”? https://education.seattlepi.com/hypomorphic-allele-4565.html
[10] 行话,KO的个体叫mutant,被MOs KD的个体叫morphant.
[11] 脑补的。
[12] El-Brolosy MA, Kontarakis Z, Rossi A, Kuenne C, Günther S, Fukuda N, Kikhi K, Boezio GLM, Takacs CM, Lai SL, Fukuda R, Gerri C, Giraldez AJ, Stainier DYR. Genetic compensation triggered by mutant mRNA degradation. Nature. 2019 Apr;568(7751):193-197. doi: 10.1038/s41586-019-1064-z. Epub 2019 Apr 3. PMID: 30944477; PMCID: PMC6707827.
[13] Egfl7和emilin3a用NW算法双序列比对的相似度约40%。图中MO代表Morpholino,NT和T是分别对非模板链和模板链CRIPSRi
[14] 问题在于,哪种类型的mRNA decay会造成Transcriptional adaptation,哪种又不会。显然不是所有的mRNA decay都会造成transcriptional adaptation. 用antisense oligonucleotides来KD也会引起RNaseH等对mRNA-DNA杂交链进行消化,或引起DICER和AGO介导的RNAi系统。mRNA decay的分类和触发条件有哪些呢?
[15] Eve AM, Place ES, Smith JC. Comparison of Zebrafish tmem88a mutant and morpholino knockdown phenotypes. PLoS One. 2017 Feb 13;12(2):e0172227. doi: 10.1371/journal.pone.0172227. PMID: 28192479; PMCID: PMC5305201.